
Le 26 juin 2017
Les scientifiques de Salk ont amélioré une approche classique pour cartographier les interactions entre les protéines
Les scientifiques de Salk ont amélioré une approche classique pour cartographier les interactions entre les protéines
LA JOLLA—Les scientifiques de Salk ont mis au point une nouvelle technique à haut débit pour déterminer quelles protéines d'une cellule interagissent les unes avec les autres. La cartographie de ce réseau d'interactions, ou «interactome», a été lente dans le passé car le nombre d'interactions pouvant être testées simultanément était limité. La nouvelle approche, publiée le 26 juin dans Nature Methods, permet aux chercheurs de tester des millions de relations entre des milliers de protéines en une seule expérience.
Cliquez ici pour une image haute résolution
Crédit: Salk Institute
"La puissance de cette nouvelle approche réside dans la capacité que nous avons maintenant de la mettre à l'échelle", déclare l'auteur principal Joseph Ecker, professeur et directeur du laboratoire d'analyse génomique de Salk et chercheur du Howard Hughes Medical Institute. "Ce test a le potentiel de commencer à répondre à des questions sur les interactions biologiques fondamentales que nous n'avons pas été en mesure d'aborder auparavant."
L'interactome d'une cellule, comme une carte des réseaux sociaux, permet aux scientifiques de voir qui travaille avec qui dans le monde des protéines. Cela les aide à comprendre les rôles des différentes protéines et à reconstituer les différents acteurs des voies et processus moléculaires. Si une protéine nouvellement découverte interagit avec de nombreuses autres protéines impliquées dans le métabolisme cellulaire, par exemple, les chercheurs peuvent en déduire qu'il s'agit d'un rôle probable pour la nouvelle protéine et potentiellement la cibler pour des traitements liés à un dysfonctionnement métabolique.
Jusqu'à présent, les chercheurs s'appuyaient généralement sur des tests standard à double hybride (Y2H) de levure à haut débit pour déterminer les interactions entre les protéines. Le système nécessite l'utilisation d'une seule protéine connue - connue sous le nom d '«appât» - pour dépister un pool de protéines «proies». Mais trouver toutes les interactions entre, par exemple, 1,000 1000 protéines, nécessiterait XNUMX XNUMX expériences distinctes pour dépister une fois les partenaires d'interaction de chaque appât.
"Les technologies actuelles exigent essentiellement que les interactions détectées lors du dépistage primaire soient retestées individuellement", explique Shelly Trigg, chercheuse diplômée de la NSF au Université de Californie, San Diego, dans le laboratoire Ecker, et premier auteur du nouvel article. "Cela peut ne plus être nécessaire avec la profondeur de dépistage que cette approche permet d'atteindre."
Dans leur nouvelle méthode, Ecker, Trigg et leurs collègues ont ajouté une touche au test Y2H standard pour un moyen beaucoup plus efficace de mesurer l'interactome. Les gènes de deux protéines, chacune sur son propre cercle d'ADN, sont ajoutés à la même cellule. Si les protéines d'intérêt interagissent à l'intérieur de la cellule, un gène appelé Cre est activé. Lorsqu'il est activé, Cre associe physiquement les deux cercles individuels d'ADN, associant ainsi les gènes des protéines en interaction afin que l'équipe puisse facilement les trouver par séquençage. L'équipe peut générer une bibliothèque massive de cellules de levure, chacune contenant différentes paires de protéines en introduisant des combinaisons aléatoires de gènes sur de l'ADN circulaire appelé plasmides. Lorsque les cellules sont positives pour une interaction protéique, les chercheurs peuvent utiliser le séquençage génétique pour déterminer quelles sont les deux protéines qui interagissent, en utilisant de nouvelles technologies de séquençage d'ADN à haut débit similaires à celles utilisées pour le séquençage du génome humain. De cette façon, ils ne sont plus limités à tester une protéine « appât » à la fois, mais peuvent tester les interactions entre toutes les protéines d'une bibliothèque à la fois.

Cliquez ici pour une image haute résolution.
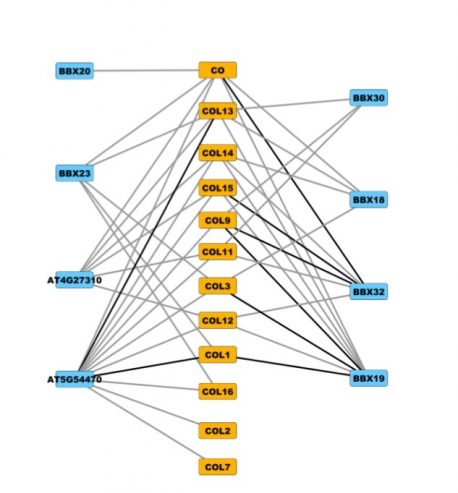
Le groupe d'Ecker a testé la nouvelle méthode, baptisée CrY2H-seq, sur tous les facteurs de transcription - une grande classe de protéines - dans la plante Arabidopsis.
"Lorsque vous prenez 1,800 4 protéines et testez les interactions entre elles, cela représente près de XNUMX millions de combinaisons", explique Ecker. "Nous avons fait cela dix fois en l'espace d'un mois."
Ils ont révélé plus de 8,000 XNUMX interactions entre ces protéines testées, leur donnant un nouvel aperçu de Arabidopsis les facteurs de transcription interagissent entre eux. Les données, disent-ils, aident à répondre à des questions de longue date sur la question de savoir si certains groupes de facteurs de transcription ont des fonctions définies. Certains des facteurs de transcription mal compris, ont-ils découvert, interagissent avec des facteurs mieux compris qui régulent la réponse de la plante à l'auxine, une hormone impliquée dans la coordination de la croissance de la plante.
À l'avenir, la méthode pourrait être mise à l'échelle pour tester de plus grands ensembles de protéines - les cellules humaines, par exemple, contiennent environ 20,000 XNUMX protéines différentes. Cette méthode plus simple et plus rapide pour déterminer l'interactome entier d'une cellule ouvre également la possibilité d'étudier comment l'interactome change dans différentes conditions - une expérience qui n'a jamais été possible dans le passé.
Les autres chercheurs de l'étude étaient Renee Garza, Andrew MacWilliams, Joseph Nery, Anna Bartlett, Rosa Castanon, Adeline Goubil, Joseph Feeney, Ronan O'Malley, Shao-shan Carol Huang, Zhuzhu Zhang et Mary Galli du Salk Institute.
Les travaux et les chercheurs impliqués ont été soutenus par des subventions du Département américain de l'énergie, Programme de bourses de recherche pour les diplômés de la Fondation nationale des sciences, Institut médical Howard Hugheset Fondation Mary K. Chapman.
BLOG
Nature Methods
AUTEURS
Shelly A Trigg, Renee M Garza, Andrew MacWilliams, Joseph R Nery, Anna Bartlett, Rosa Castanon, Adeline Goubil, Joseph Feeney, Ronan O'Malley, Shao-shan C Huang, Zhuzhu Z Zhang, Mary Galli et Joseph R Ecker
COUVERTURE DU JOURNAL
Une méthode à double hybride de levure massivement multiplexée, CrY2H-seq, permet une cartographie en profondeur de la Arabidopsis interactome. Couverture préparée par Erin Dewalt, basée sur la conception et les illustrations de Shelly Trigg, Lisa Servilio et Jamie Simon au Salk Institute for Biological Studies et par Austin Trigg à Austin Trigg Photography.
Bureau des communications
Tél: (858) 453-4100
presse@salk.edu
Percer les secrets de la vie elle-même est la force motrice du Salk Institute. Notre équipe de scientifiques primés de classe mondiale repousse les limites des connaissances dans des domaines tels que les neurosciences, la recherche sur le cancer, le vieillissement, l'immunobiologie, la biologie végétale, la biologie computationnelle et bien plus encore. Fondé par Jonas Salk, développeur du premier vaccin sûr et efficace contre la polio, l'Institut est un organisme de recherche indépendant à but non lucratif et un monument architectural : petit par choix, intime par nature et intrépide face à tout défi.